小艺
【科学背景】
聚氨酯作为全球年消耗量达2200万吨的第二大可水解塑料,其生产过程能源密集且温室气体排放高(331 MJ/kg能耗,12.3 kg-CO₂e/kg排放),但回收面临严峻挑战:热固性聚氨酯的交联结构和化学稳定的氨基甲酸酯键阻碍了酶解过程。尽管化学-酶法回收显示出潜力,现有已知的urethanases在工业糖酵解条件下效率低下,尤其对占市场75%的聚醚型聚氨酯缺乏有效降解酶。此外,糖酵解产生的底相(占产物60%以上)富含N-芳基氨基甲酸酯和过量二甘醇,目前多作为危险废物焚烧,造成环境和经济负担。因此,开发高效、耐有机溶酶的urethanases成为突破聚氨酯回收瓶颈的关键。

【创新成果】
10月30日,北京化工大学(中国科学院微生物研究所)吴边教授和崔颖璐副研究员等研究者在Science上发表了题为“Glycolysis-compatible urethanases for polyurethane recycling”的论文,提出了GRASE(基于图神经网络的活性与稳定性酶推荐)框架,通过整合自监督和监督学习,从蛋白质数据库中筛选高效且兼容糖酵解条件的urethanases。其中,ADPURase在6摩尔二甘醇中活性比已知酶提高两个数量级,可在8小时内实现千克级商业聚氨酯近乎完全解聚。结构分析表明,其紧密的疏水核心和脯氨酸稳定的盖状结构域赋予了在苛刻溶剂中的稳定性与效率。这项工作展示了深度学习在加速具有工业潜力生物催化剂发现方面的优势,并为聚氨酯回收提供了可行解决方案。
【数据概览】
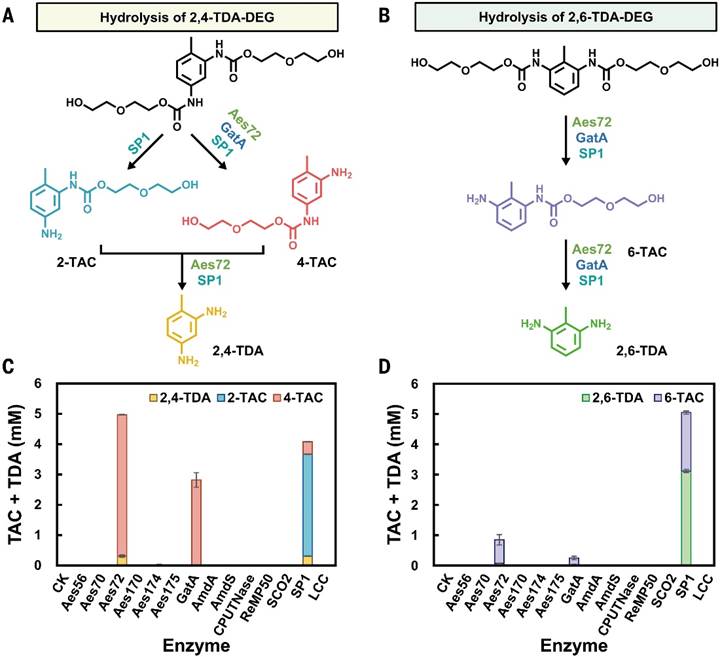
图1 TDA-DEG的水解途径和文献中声称的PURases的酶活性© 2025 AAAS
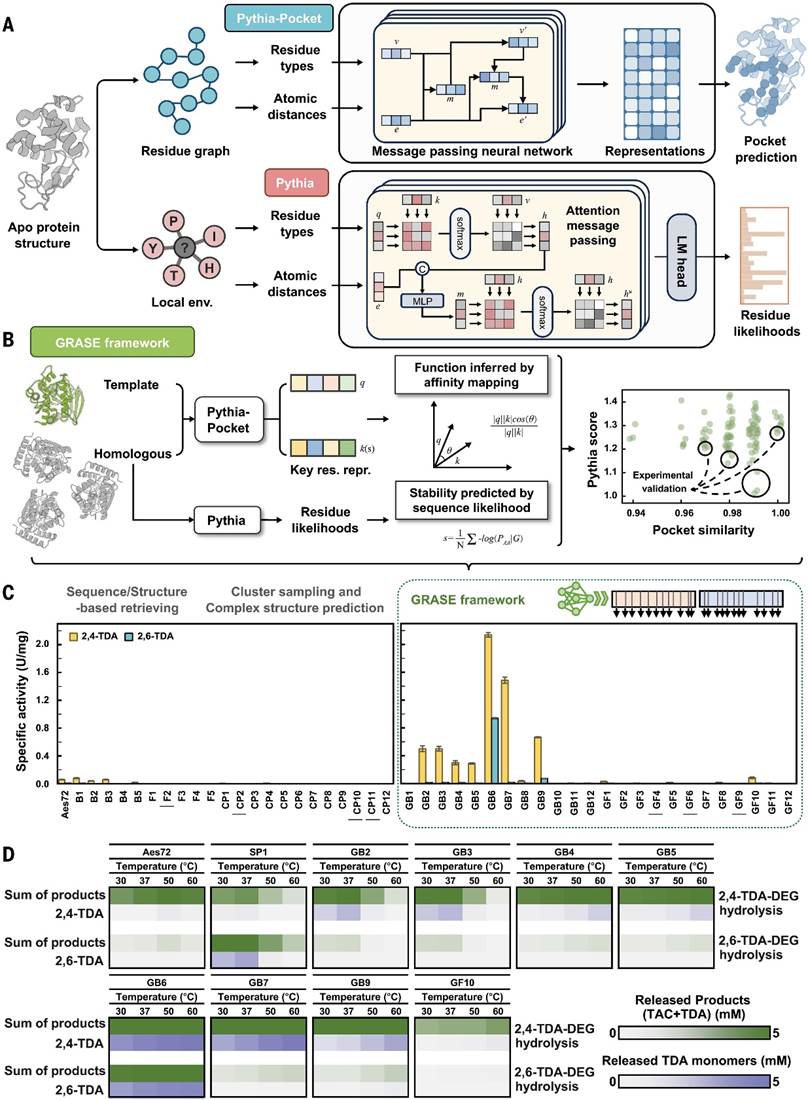
图2 GRASE模型架构及其鉴定酶的功能验证结果© 2025 AAAS
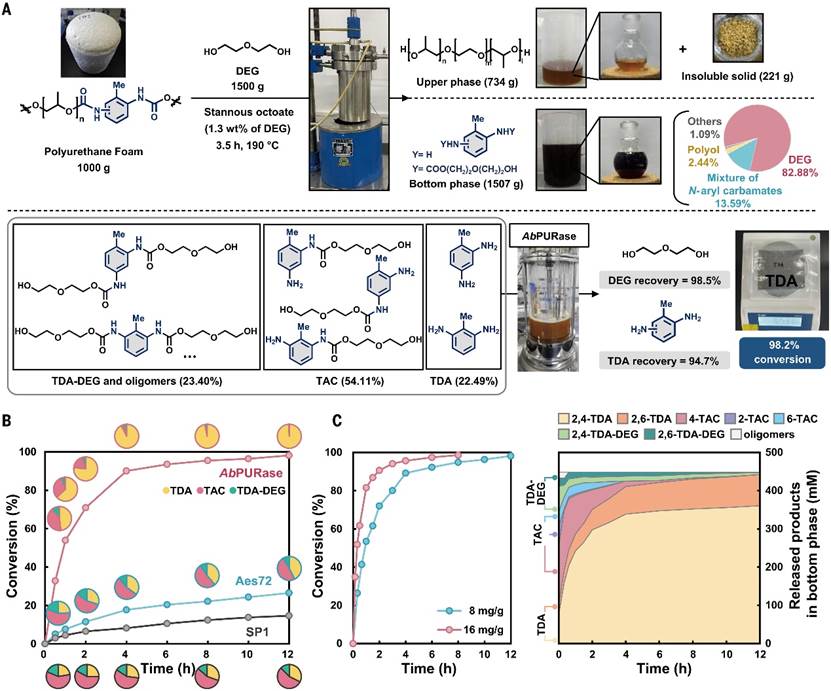
图3 BASF聚氨酯泡沫的化学-酶法解聚过程及千克级规模应用© 2025 AAAS
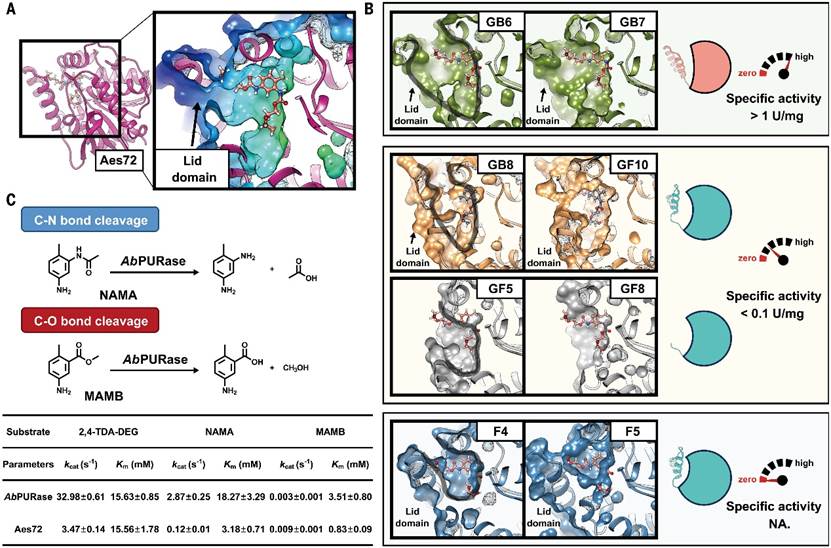
图4 不同酶的结构特征及动力学参数比较© 2025 AAAS
【科学启迪】
研究显示,基于表征的学习策略(如GRASE框架)能更可靠地推断酶功能,尤其通过局部活性位点嵌入识别远缘同源酶(序列一致性低至34%)。ADPURase虽被传统归类为酯酶,但实际主要发挥urethanase功能,体现了酶的多功能性和“月光效应”。未来研究应聚焦于捕捉和放大这些隐藏功能信号,以深化对酶功能库的理解,并开发适用于多种工业挑战的生物催化剂。此外,需通过蛋白质工程进一步提升酶性能,优化回收单体再聚合过程,推动规模化应用。
文献链接:Yanchun Chen et al., Glycolysis-compatible urethanases for polyurethane recycling. Science 390, 503-509 (2025). DOI:10.1126/science.adw4487